W periodyku Agriculture Ecosystems & Environment ukazał się artykuł zespołu prof. Arkadiusza Nowaka na temat podziemnej bioróżnorodności (5.567, 200 pkt).
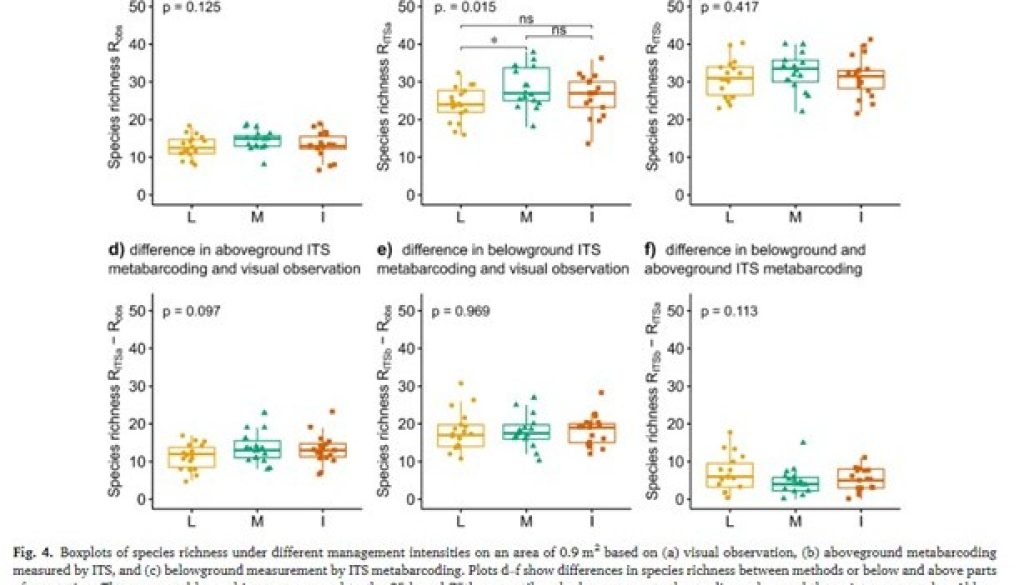
Wykorzystując metabarkodowanie eDNA po raz pierwszy rozpoznano proporcje między nadziemnymi i podziemnymi częściami roślinności w trzech typach traworośli. Przeanalizowano także czynniki takie jak wilgotność, żyzność i intensywność użytkowania na kompozycję gatunkową łąk. Metabarkodowanie eDNA z powodzeniem przypisało około 93% taksonów na poziomie gatunkowym. Nasze badania ujawniły, że pod ziemią zbiorowisko roślinne składa się do 66% większej liczby gatunków w porównaniu do nadziemnej części zidentyfikowanej za pomocą konwencjonalnych badań roślinności. Jednak różnica ta zmniejsza się do około 25%, gdy przypisanie taksonomiczne pochodzące z eDNA zostało zastosowane także dla nadziemnej części fitocenozy. Stwierdziliśmy również, że różnica między pod- i nadziemną częścią zbiorowiska roślinnego zmniejszała się wraz ze wzrostem żyzności gleby. Ustaliliśmy, że intensywność gospodarowania istotnie wpływa na bogactwo gatunkowe tylko w nadziemnej części roślinności, przy czym podziemna część użytków zielonych pozostaje zachowana.
Rucińska A., Świerszcz S., Nobis M., Zubek S., Boczkowska M., Olszak M., Kosiński J.G., Nowak S., Nowak A. 2021. Is it possible to understand a book missing a quarter of the letters? Unveiling the belowground species richness of grasslands. Agriculture Ecosystems & Environment 324:107683
DOI: 10.1016/j.agee.2021.107683
Abstract: Knowledge of species richness patterns in plant communities is biased toward its hidden belowground part and is derived only from the part visible aboveground. Studies on the relationship of the above- to belowground parts of plant communities suffer from the lack of a consistent and uniform approach to assess their taxonomic composition, particularly in species-rich ecosystems. This study described the proportion between above- and belowground parts of vegetation in three grassland types along with the environmental factors that affect them, using eDNA metabarcoding and an observer survey. The internal transcribed spacer 2 (ITS2) region was used to analyze the total plant richness in the soil and the aboveground “green” part of plant communities. Considering all samples, eDNA metabarcoding successfully assigned about 93% of taxa on the species level. Our findings support the notion that metabarcoding analysis of the belowground plant community revealed up to 66% higher species richness than was identified above the ground by a conventional vegetation survey. However, this difference decreases to about 25% when an eDNA-derived taxonomic assignment was applied for the aboveground part of phytocoenosis. We also found that the difference between the below- and aboveground parts of the plant community decreased with increased soil fertility. Finally, the management intensity was found to significantly influence species richness only in the aboveground part of vegetation with the belowground part of grassland remaining unaffected. Overall, since DNA-based metabarcoding and traditional vegetation surveys have limitations, their complementary use is recommended to obtain the most reliable vegetation description. However, it should be considered that metabarcoding analysis is destructive and may not be applicable in protected or private areas or on permanent research plots.